

Plateforme: Computational Biology, Biostatistics & Modeling (CB2M)
À propos
Le CB2M (Computational Biology, Biostatistics & Modeling) est la plateforme de biologie computationnelle du CIML dédiée à l’avancement de la recherche en immunologie au CIML grâce à la biologie computationnelle. Il offre une expertise dans plusieurs domaines, notamment le développement de méthodes et d’outils computationnels, l’analyse de données biologiques, les statistiques et la modélisation physique et mathématique.
En créant des liens interdisciplinaires entre les immunologistes et les disciplines de la biologie computationnelle, le CB2M vise à apporter les approches les plus innovantes en matière de biologie computationnelle au CIML.
Pour relever ces défis complexes, le CB2M fonctionne selon deux approches stratégiques complémentaires :
1. Un groupe de scientifiques experts
La biologie computationnelle est devenue un pilier fondamental de la recherche immunologique contemporaine. À mesure que la technologie progresse, les méthodes d’analyse des données deviennent de plus en plus sophistiquées, intégrant des concepts mathématiques et statistiques d’une complexité sans précédent. Ces approches algorithmiques permettent désormais d’explorer des dimensions immunitaires jusqu’alors inaccessibles, transformant ainsi notre compréhension du système immunitaire. Cependant, l’utilisation d’outils computationnels modernes pour la biologie nécessite une combinaison unique de compétences.
Les scientifiques du CB2M apportent au CIML ces compétences :
- en analyse de données et statistiques, grâce à leur expertise en bio-informatique et en mathématiques pures et appliquées ;
- en modélisation mathématique et physique des systèmes naturels, grâce à leur formation initiale en physique théorique ;
- en informatique théorique et génie logiciel, grâce à leur formation et leur expérience professionnelle ;
- en relations interdisciplinaires, grâce à leurs interactions passées avec de nombreux biologistes.
2. Un écosystème coopératif
Les projets de recherche menés au CIML nécessitent des analyses computationnelles biologiques approfondies, complexes et de longue haleine. Ces analyses font appel à des technologies de plus en plus avancées (par exemple, l’omique unicellulaire, l’omique spatiale) qui sont de plus en plus souvent associées à la cytométrie, à l’imagerie et à l’histologie. Le CB2M s’engage à aider les chercheurs à relever ces défis et à leur permettre de produire des travaux scientifiques de la plus haute qualité.
Le recrutement d’un ou plusieurs bioinformaticiens spécialement dédiés à leurs projets est le moyen le plus efficace pour les équipes du CIML de réaliser des analyses robustes en biologie computationnelle. Cependant, pour la plupart des chercheurs principaux en biologie, le recrutement et la supervision de bioinformaticiens constituent un défi. De plus, les bioinformaticiens isolés manquent d’occasions d’interagir avec des experts dans leur domaine afin d’adopter les meilleures pratiques analytiques.
Au CIML, le CB2M offre un environnement coopératif où biologistes et bioinformaticiens peuvent facilement interagir. Ici, les bioinformaticiens sont entourés de collègues de leur domaine et soutenus par des experts, mais ils conservent des liens étroits avec leur équipe d’origine. Au sein de son écosystème collaboratif, le CB2M fournit son expertise, des formations et un encadrement à tous les bioinformaticiens du CIML.
Formation
Le CB2M s’engage à former le personnel du CIML aux connaissances, méthodes et techniques associées à la biologie computationnelle et aux statistiques. Le CB2M a développé un programme de formation complet destiné aux bioinformaticiens et aux biologistes souhaitant acquérir davantage de connaissances et de compétences en biologie computationnelle.
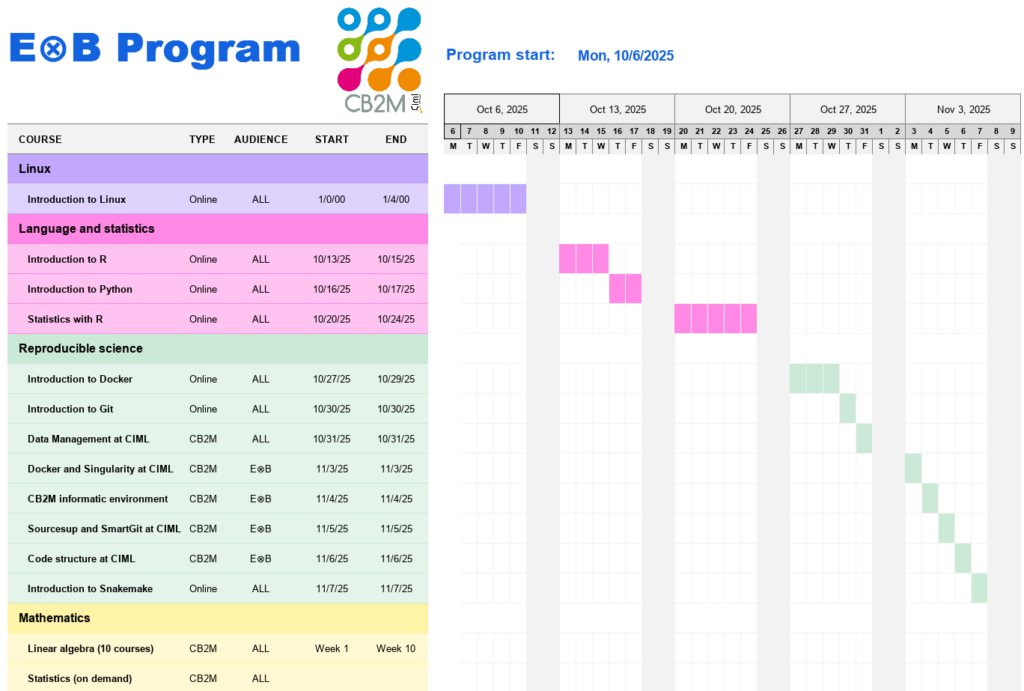
Ce programme, appelé Programme E⊗B (Experimentalist Cross Bioinformatician Program), comprend les modules suivants (les détails des modules sont présentés dans le diagramme de Gantt ci-dessous) :
- Linux : ce module vise à fournir des connaissances et des pratiques de base sur le système d’exploitation Linux.
- Langage et statistiques : ce module présente les langages R et Python ainsi que l’application des statistiques de base pour l’analyse des données.
- Science reproductible : ce module propose des cours visant à fournir des méthodologies, des outils et des bonnes pratiques pour garantir la reproductibilité des analyses bioinformatiques. Ce domaine est le tronc commun à tous les cours.
- Mathématiques : ce module vise à enseigner ou à approfondir les connaissances mathématiques les plus couramment utilisées dans les analyses effectuées au CIML.
Le programme est adapté aux besoins de chaque individu :
- Pour les bioinformaticiens (stagiaires, doctorants, ingénieurs, post-doctorants), les cours sont obligatoires et sont sélectionnés en fonction du niveau de l’apprenant.
- Pour les biologistes qui souhaitent acquérir une double compétence en biologie et en bioinformatique (principalement des doctorants), il est proposé de suivre le programme E⊗B complet avec un planning adapté à leurs objectifs.
Veuillez noter qu’en plus du programme E⊗B :
- Le CB2M propose des cours sur des sujets spécifiques au personnel du CIML, tels que l’analyse des données RNA-seq unicellulaires à l’aide de l’interface ShIVA ou les statistiques. Ces cours sont annoncés longtemps à l’avance et sont soumis à une inscription volontaire.
- Les membres du CB2M sont également formateurs pour des cours externes, tels que les formations de l’Inserm. Les membres du CIML sont invités à s’inscrire à ces cours si le sujet les intéresse.
Voici un exemple de planning du Programme E⊗B:
Open Science
Le CB2M est fortement engagé en faveur de la science ouverte et de la reproductibilité scientifique. Afin de garantir la fiabilité et la transparence des résultats de recherche, le CB2M a mis en place des bonnes pratiques et des méthodes permettant d’assurer la reproductibilité à 100 % de toutes les analyses bioinformatiques réalisées dans le cadre des projets scientifiques.
Un élément essentiel de cette mission est la formation : chaque bioinformaticien recruté au CIML, ainsi que les étudiants accueillis en stage, est formé à ces pratiques et accompagné afin de s’assurer de leur bonne application. Cette démarche contribue à instaurer une véritable culture de rigueur et de reproductibilité dans l’ensemble des activités de biologie computationnelle du CIML.
Le CB2M joue également un rôle actif dans l’adoption des infrastructures nationales pour la science ouverte. Il accompagne ainsi l’utilisation de Recherche Data Gouv, la plateforme officielle française de stockage des données scientifiques. Grâce à l’implication du CB2M, le CIML fait partie des premiers laboratoires d’Aix-Marseille Université à disposer de son propre espace dédié au sein de l’entrepot de Recherche Data Gouv. Le CB2M participe également aux Assises Nationales sur les Données de la Recherche (ANDOR).
À travers ces actions, le CB2M renforce l’ouverture, la transparence et l’impact à long terme des recherches en immunologie menées au CIML.
Projets
Project : DECITIP Les cellules dendritiques plasmacytoides (pDCs) excellent dans la production des interférons de type I et III (IFN), […]


Notre santé repose sur un équilibre délicat entre la défense immunitaire contre les agents pathogènes et la tolérance vis à […]


En collaboration avec Denis Puthier (TAGC) and Lionel Spinelli (CB2M, CIML), nous avons co-développé SciGeneX, un package R innovant permettant […]

Lymph nodes are formed in the embryo by Lymphoid Tissue Inducer (LTi) cells. LTi cells arise not from the yolk […]

Innate immune cells are essential partners of the central nervous system, supporting both homeostasis and repair. Within the brain, perivascular […]

Projet : Treating Breast Cancer Le cancer du sein est le cancer le plus fréquemment diagnostiqué chez les femmes dans […]

Projet : TREATLIVMETS Les métastases hépatiques surviennent chez près de 50 % des patients atteints de cancer, en particulier dans […]

Project : pDCComp Les individus infectés par des virus ou souffrant de maladies inflammatoires développent un ensemble de troubles comportementaux […]

Project : PLBIO 2024 Coordinateur: Thierry Walzer, CIRI, Lyon. Autre participant: Nathalie Bendriss-Vermare, CRCL, Lyon. Résumé: Les cellules Natural Killer […]

Project : ACLAMADABREC En partie via leur efficacité à activer les cellules natural killer (NK) et les lymphocyte T CD8 […]

Project : MITI Autres participants: Magali Richard, LIG, Grenoble; Lionel Spinelli, hub CB2M du CIML. Résumé: Ce projet vise à […]

Project : UNFIRE Autres participants: Ciphe (Ana Zarubica), Hospices Civils de Lyon et CIRI (Sophie Trouillet-Assant, Thierry Walzer), équipe CoVir […]





Project : PLBIO-25 Coordinateur: Marianne BURBAGE, Institut Curie, Paris. Autre participant: Andrew GRIFFITHS, ESPCI, Paris. L’efficacité des lymphocytes T CD8 […]

Project : DCFUNDEVSCREEN Résumé: Des études chez la souris ont montré le rôle clé antiviral/tumoral des cellules dendritiques, plasmacytoïdes (pDCs) […]

Project : RIPIREVI Autres participants: CIPHE (Ana Zarubica et collègues). Résumé: Les interférons de type I (IFN-Is) sont essentiels à […]


Project : LYMPHOMATLAS Des études multi-omiques en cellule unique dans les lymphomes B ont permis de mettre en lumière un […]


Project : NRegSkin Porteur: Mei LI (IGBMC, Strasbourg). Autres participants: Katia Boniface, Julien Seneschal (Université de Bordeaux). Résumé: La peau […]

La progression du lymphome folliculaire est marquée par des cycles iteratifs de rémission et de rechute, ces dernières résultant généralement de la […]

Project : REVOLUTION Coordinateur: Edouard Sage, Hôpital Foch, Suresnes. Autres partenaires: Isabelle Schwartz-Cornil, INRAE, Jouy-en-Josas; Jérémie Pourchez, Ecole des Mines […]

Project : ICARO Les vaccins acellulaires actuels sont surtout efficaces contre les pathogènes extracellulaires, mais échouent à induire une réponse […]


Project : DeCoDis In the context of a European consortium, we are investigating the dynamics and function of border-associated macrophages […]

Afin d’identifier de nouvelles combinaisons thérapeutiques et comprendre les mécanismes de résistance aux therapies ciblées dans les hémopathies lymphoïdes derives […]

Les patients atteints de lymphome folliculaire (LF) présentent une très grande hétérogénéité biologique impactant fortement la réponse aux thérapies actuelles. […]

Nous avons démontré que la neurokine TAFA4, produite par des neurones sensoriels de la peau, peut promouvoir la réparation tissulaire […]

La réponse de l’hôte aux pathogènes est orchestrée par le système immunitaire et le système nerveux, tous deux présents dans […]

Au delà des processus de perception de la douleur qui sont induits en cas de lésion ou d’infection de la […]

Le projet TCELL-CODE, coordonné par le Dr Rémy Lasserre au Centre de Recherche en Cancérologie de Marseille vise à élucider […]


L’action structurante Spatial Omics du consortium CALYM, coordonnée par le Dr Pierre Milpied, vise à développer une plateforme virtuelle dédiée […]

Le projet GCselection, coordonné par le Dr Pierre Milpied (CIML), revisite les mécanismes de maturation des anticorps dans les centres […]


L’action structurante ST-omics du Cancéropôle de la Région Sud est co-coordonnée par le Dr Pierre Milpied (CIML), le Pr Emmanuelle […]

Le projet AITLas-Impact, coordonné par Pierre Milpied (CIML) en collaboration avec le Pr François Lemonnier (Institut Mondor de Recherche Biomédicale, […]

L’équipe du Dr Pierre Milpied au CIML participe à un projet collaboratif coordonné par le Dr Jean-Pierre de Villartay (Institut […]


